

岭南现代临床外科 ›› 2020, Vol. 20 ›› Issue (02): 167-171.DOI: 10.3969/j.issn.1009-976X.2020.02.007
摘要:
结直肠癌肿瘤是目前我国高发的恶性肿瘤之一[1]。深入研究结直肠癌的基因分子结构变化机制,寻找新的结直肠癌治疗手段和方法是目前肿瘤研究的一个热点。肿瘤侵袭和细胞转移是可能导致结直肠患者死亡的主要危险原因[2]。胶原蛋白家族是构成基底膜的重要结构性成分[3]。近来研究报告中发现,IV型结直肠癌胶原蛋白家族α1(collagentypeⅳalpha1,COL4A1)在胃癌、乳腺癌、膀胱癌等恶性肿瘤中异常地表达,并与肿瘤的发生及其发展、转移等密切相关[4-6]。但目前国外有关COL4A1基因在结直肠癌发生发展中的作用的研究较少。本研究主要采用了挖掘公开的结直肠癌生物化学数据库的研究方法,利用Oncomine数据库[7]、GEPIA数据库[8]、MethHC甲基化数据库[9]和String在线公开数据库[10]等综合分析了COL4A1基因在结直肠癌中的表达和预后情况,分析其可能参与的机制,为进一步COL4A1基因在结直肠癌中的作用提供理论依据。
Oncomine基因芯片数据库平台是一个肿瘤基因芯片筛选数据库和基因整合数据库收集和挖掘的平台,注册后用户可在基因芯片数据库中根据用户需求自动设定关键筛选的条件。本研究首先将筛选条件设定为:gene:COL4A1,analysis type:cancer vs.normal analysis,data type:mRNA。通过Oncomine在线公开数据库分析COL4A1基因在不同人类肿瘤中的表达情况。然后增加筛选条件:cancer type:coloncarcinoma,threshold by P<0.05,foldchange:all,generank:all
GEPIA数据库(http://gepia.cancer-pku.cn/)是由北京大学研制开发的用于整合分析癌症表达谱数据的交互式web服务器,包含了TCGA数据库和GTEx数据库的33种恶性肿瘤,共有9736个肿瘤样本和8587个正常样本的RNA测序表达数据。在本研究中,我们应用GEPIA数据库验证之前应用Oncomine数据库所得出的相关结果,并用GEPIA数据库中包含TCGA数据库的数据,分析COL4A1基因的表达水平与病理分级、生存预后的关系。
MethHC数据库整合了TCGA数据库中人类肿瘤基因组的甲基化数据,基于此分析COL4A1基因甲基化与结肠腺癌的相关性。检索条件限定如下:①search by:gene;②cancer:COAD&READ;③Select a gene region:Promoter;④select a methylationlevelevalution method:Average;⑤ Gene:COL4A1.
应用String蛋白网络(https://string-db.org/)分析可预测蛋白质与蛋白质相互调控网络,通过String数据库预测COL4A1蛋白的上下游调控蛋白及蛋白间的相关调控关系。本研究中输入检索条件:①Protein Name:COL4A1;②物种类型:homo sapiens。
在Oncomine数据库中共检索得到441个不同的类型恶性肿瘤的统计结果(参见图1A),其中大约有74个统计结果显示COL4A1高表达,原发性脑肿瘤、中枢神经系统恶性肿瘤、乳腺癌、宫颈癌、结直肠癌、食管癌、胃癌、头颈部乳腺癌、肾癌、肝癌、肺癌、淋巴瘤、胰腺癌、卵巢癌、肉瘤及其他恶性肿瘤等;5个临床检查结果显示其恶性肿瘤可能低表达,且主要表现集中在包括膀胱癌、肺癌、黑素瘤、肉瘤等原发性癌症。
在Oncomine数据库中对描述结肠腺癌的有关研究进行分析,共有11项符合条件的研究,分别对符合条件的11项研究进行对比分析,发现每一项研究中结肠腺癌组织中COL4A1基因表达均高于正常对照组织。为了进一步明确两者的相关性,对11项研究进行荟萃分析,结果显示结直肠癌组织中COL4A1基因mRNA的表达水平明显高于正常组织(P=1.29E-5),见图1B。其中有六项结直肠癌研究结果提示,其表达差异具有显著性(P<0.05)。通过GEPIA数据库,分别在结肠癌与直肠癌组织中进一步验证COL4A1转录水平较正常结肠和直肠组织表达量要明显增高(P<0.05,图2A)。
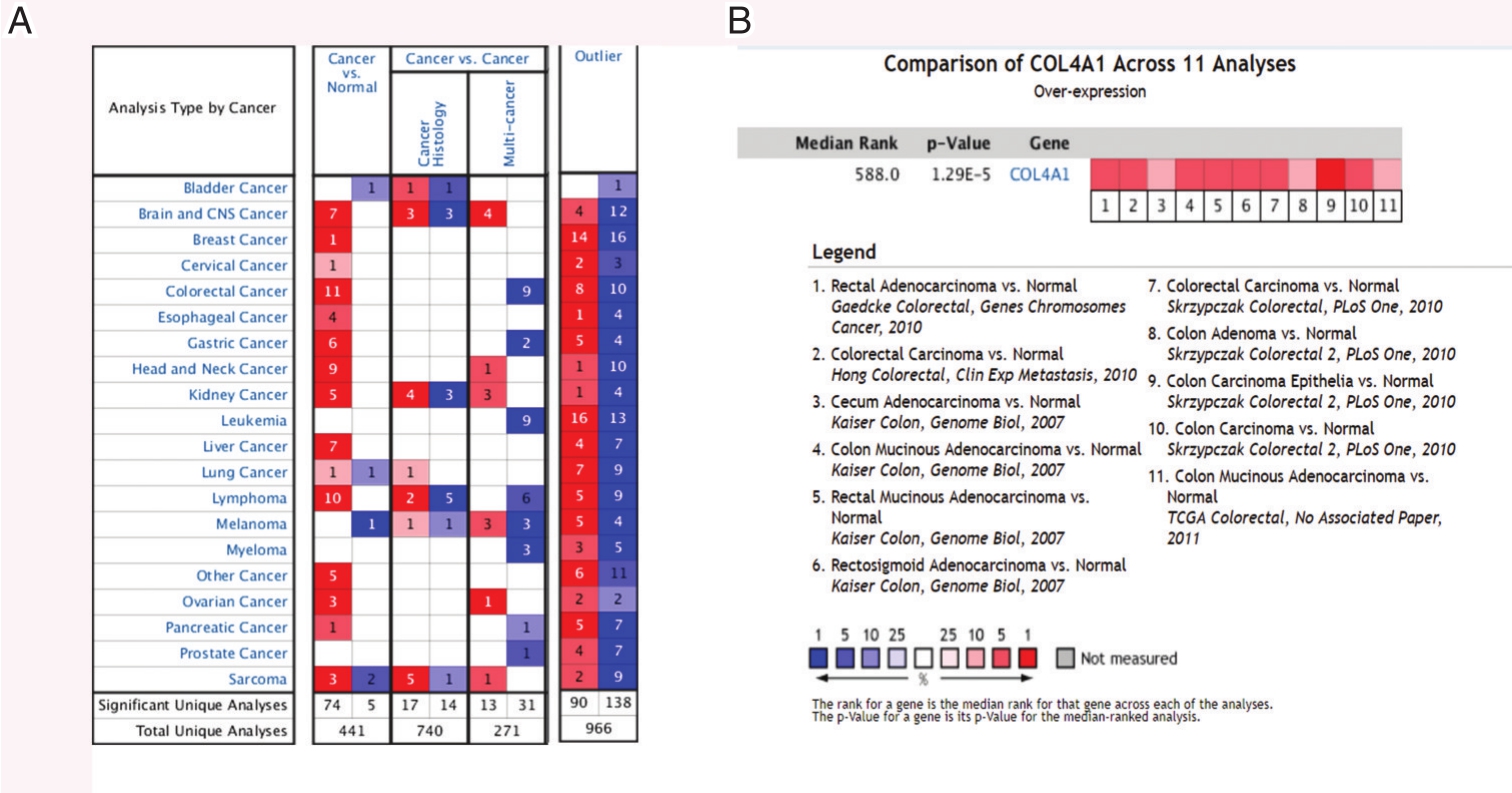
图1 Oncomine数据库中COL4A1基因的表达情况
A.COL4A1在不同肿瘤中的差异性表达;B.Oncomine数据库中11项研究荟萃分析显示COL4A1基因在结直肠癌组织中的表达差异
在GEPIA数据库中,我们分析了COL4A1基因表达与结肠癌和直肠癌的临床病理分期的相互关系,发现无论是结肠癌还是直肠癌,在不同的肿瘤临床分期各组间COL4A1 mRNA表达差异均无统计学意义(结肠癌组F=2.3,P=0.0773;直肠癌组F=2.06,P=0.112;见图2B)。我们再利用GEPIA数据库进行生存分析发现COL4A1基因的表达与结直肠癌的生存率和预后无关(P>0.05)。
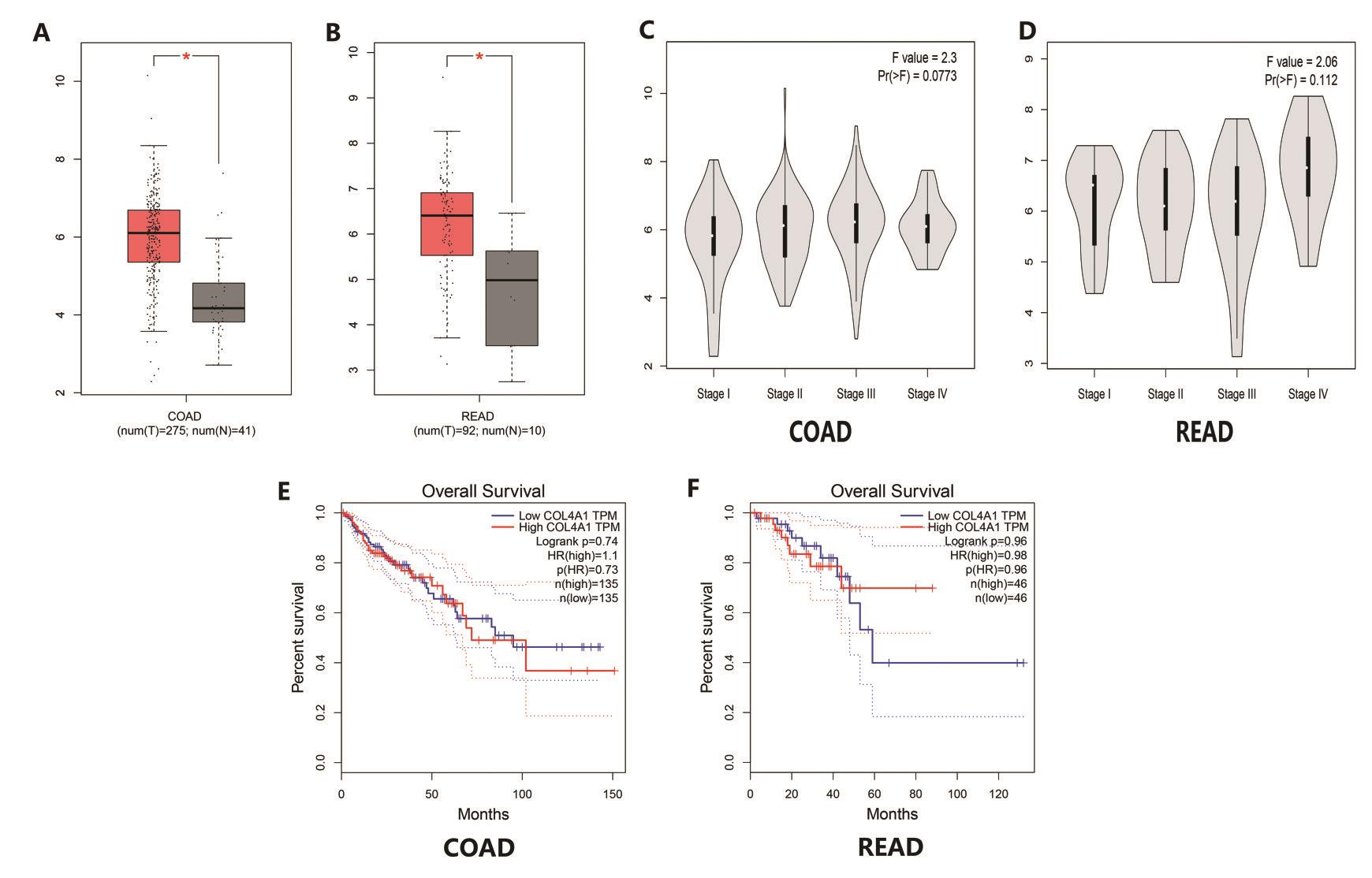
图2 GEPIA数据库中COL4A1在结直肠癌中的表达、临床病理分期及预后的关系
A.COL4A1在结直肠癌中的表达;B.COL4A1在直肠癌中的表达;C.COL4A1与结肠癌临床病理分期的关系;D.COL4A1与直肠癌临床病理分期的关系;E.C.COL4A1与结肠癌生存预后的关系;F.C.COL4A1与直肠癌临床病理分期的关系。COAD:结肠腺癌;READ:直肠腺癌
在MethHC数据库中,检索发现在结直肠癌组织中COL4A1基因有一个转录模板NM_001845。分析结果发现,COL4A1在结肠癌和直肠癌组织中甲基化水平明显升高(P<0.005)。甲基化数据和表达谱数据表达量的相关性分析提示:COL4A1在结直肠癌中的表达与甲基化正相关,甲基化数据和表达谱数据差异倍数的相关性分析提示P<0.01。见图3A、3B。
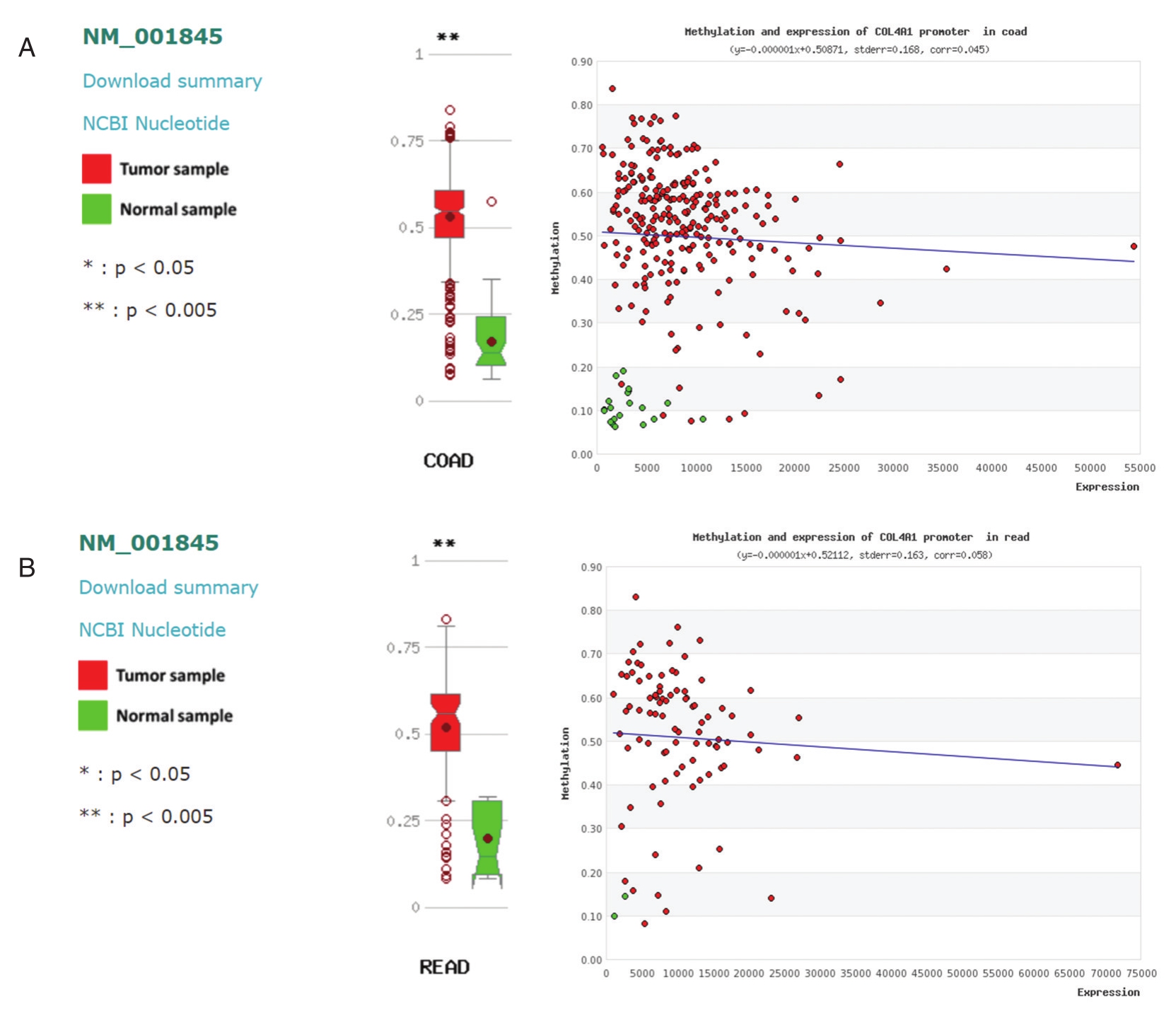
图3 MethHC数据库中COL4A1在结直肠癌与正常组织中的甲基化水平,及COL4A1在结直肠癌中的表达水平与甲基化水平的关系 COAD:结肠腺癌;READ:直肠腺癌
通过STRING分析获得COL4A1相关蛋白作用网络图,见图4。COL4A1主要相关蛋白分别为 COL16A1、 SERPINH1、 COL5A2、 COL6A1、COL5A2、COL5A1、COL1A1、COL3A1、COL4A2、COL1A2、ITGA1,且相互作用 score>0.95。将以上相关蛋白进行富集分析,发现这些基因主要参与了细胞外基质组织,胶原蛋白纤维组织,细胞对氨基酸刺激的反应,对酸性化学品的反应,蛋白质异三聚化等生理过程。而信号通路分析预测COL4A1基因可能在ECM受体相互作用、局部粘附等信号通路中发挥作用。

图4 COL4A1基因与上下游基因的调控关系
肿瘤细胞的侵袭转移与细胞外基质(extracellular matrix,ECM)密切相关[11]。细胞基底膜是由COL4A1蛋白、层粘连蛋白和纤连蛋白构成的一种特殊形式的ECM(12)。基底膜的主成分是COL4A1蛋白。已有研究表明COL4A1基因在乳腺癌中高表达,且在乳腺癌细胞的增殖和集落形成中发挥关键作用[4]。尿路上皮癌细胞中COL4A1基因亦为高表达,且高表达COL4A1基因是其复发的独立危险因素[6]。COL4A1基因同样在胃癌组织中高表达,COL4Al蛋白可能与胃癌细胞ECM受体相互作用,参与胃癌细胞的转移和侵袭[5]。但目前有关COL4Al在结直肠癌中的表达、预后及可能涉及的机制等研究较少。
为了探索COL4A1基因在结直肠癌中的作用,本研究首先应用Oncomine在线数据库,挖掘COL4A1基因在常见肿瘤中的表达情况。结果显示,COL4A1在肿瘤组织中高表达的结果有74个,其中有11项研究显示,COL4A1基因在结直肠癌组织中高表达。荟萃分析显示,总体上COL4A1基因在结直肠癌组织中的mRNA表达水平高于正常结直肠黏膜组织。有六项结直肠癌研究表明,COL4A1基因mRNA表达水平显著升高,差异具有显著性。我们通过GEPIA数据库分析验证了Oncomine数据库的结果。据此推测,COL4A1基因在结直肠癌的发生过程中可能扮演重要的角色。但随后的临床病理分期及预后结果提示,差异无统计学意义。分析可能原因,其一,上述研究结论仅仅是在转录水平上得出的,有一定局限性;其二,肿瘤的分期与预后受到多因素共同参与调节,绝非某一个促癌因子能起到决定性作用,COL4A1的作用可能被其他拮抗因子影响。后续研究中,通过进一步验证COL4A1在蛋白水平对结直肠癌生物学行为的影响,探索参与COL4A1的上游调控与修饰的关键分子具有重要的科学研究价值。因此我们推测,COL4A1可能在发生是过程中可能是个关键因子。
为了研究影响COL4A1基因在结直肠癌组织中表达差异的可能机制,我们通过MethHC在线数据库分析了结直肠癌组织和正常结直肠黏膜组织中COL4A1基因DNA启动子区的甲基化水平的变化,结果显示:结直肠癌组织中COL4A1基因在DNA启动子区的甲基化水平显著升高,且甲基化水平与COL4A1的表达水平呈正相关(P<0.01),这一发现有待于进一步实验验证和探讨。另外,我们还通过String-DB数据库分析了COLA41可能参与的信号通路及相关作用蛋白,结果发现:COL4A1蛋白可能在ECM受体相互作用、局部粘附等信号通路中发挥作用。以上结果进一步提示:COL4A1可能是结直肠癌发生发展中的关键枢纽基因,通过与ECM受体相互作用,影响着肿瘤细胞的浸润、转移等生物学行为。
综上所述,通过研究分析,COL4A1基因可能参与了结直肠癌的发生发展,这些结果需进一步研究证实。
[1] Chen W,Zheng R,Baade PD,et al.Cancer statistics in China,2015[J].CA Cancer J Clin,2016,66(2):115-132.
[2] Najafi M,Farhood B,Mortezaee K.Extracellular matrix(ECM)stiffness and degradation as cancer drivers[J].J Cell Biochem,2019,120(3):2782-2790.
[3] Xia C,Lin S,Yang J,et al.Genetic Variations of COL4A1 Gene and Intracerebral Hemorrhage Outcome:A Cohort Study in a Chinese Han Population [J].World Neurosurg,2018,113:e521-e528.
[4] Jin R,Shen J,Zhang T,et al.The highly expressed COL4A1 genes contributes to the proliferation and migration of the invasive ductal carcinomas[J].Oncotarget,2017,8(35):58172-58183.
[5] Huang R,Gu W,Sun B,Gao L.Identification of COL4A1 as a potential gene conferring trastuzumab resistance in gastric cancer based on bioinformatics analysis[J].Mol Med Rep,2018,17(5):6387-696.
[6] Miyake M,Hori S,Morizawa Y,et al.Collagen type IV alpha 1(COL4A1)and collagen type XIII alpha 1(COL13A1)produced in cancer cells promote tumor budding at the invasion front in human urothelial carcinoma of the bladder[J].Oncotarget,2017,8(22):36099-36114.
[7] Rhodes DR,Kalyana-Sundaram S,Mahavisno V,et al.Oncomine 3.0:genes,pathways,and networks in a collection of 18,000 cancer gene expression profiles[J].Neoplasia,2007,9(2):166-180.
[8] Tang Z,Li C,Kang B,et al.GEPIA:a web server for cancer and normal gene expression profiling and interactive analyses[J].Nucleic Acids Res,2017,45(W1):W98-W102.
[9] Huang WY,Hsu SD,Huang HY,et al.MethHC:a database of DNA methylation and gene expression in human cancer[J].Nucleic Acids Res,2015,43(Database issue):D856-861.
[10] Szklarczyk D,Morris JH,Cook H,et al.The STRING database in 2017:quality-controlled protein-protein association networks,made broadly accessible[J].Nucleic Acids Res,2017,45(D1):D362-D368.
[11] Wei B,Zhou X,Liang C,et al.Human colorectal cancer progression correlates with LOX-induced ECM stiffening[J].Int J Biol Sci,2017,13(11):1450-1457.
[12] Hoshiba T.An extracellular matrix(ECM)model at high malignant colorectal tumor increases chondroitin sulfate chains to promote epithelial-mesenchymal transition and chemoresistance acquisition[J].Exp Cell Res,2018,370(2):571-578.
Analysis of COL4A1 gene expression and significance in colorectal cancer based on database mining
中图分类号: