

岭南现代临床外科 ›› 2019, Vol. 19 ›› Issue (06): 755-759.DOI: 10.3969/j.issn.1009-976X.2019.06.025
摘要:
随着临床研究的深入,免疫系统在脓毒症发病过程中的认识得到不断提高,治疗的重点在于重建机体的免疫平衡状态,方法涵盖了多个方面:抑制固有免疫,清除内毒素,增强调节免疫等[1-3]。然而,脓毒症免疫治疗相关的临床研究结论并不一致,潜在有效的免疫治疗药物在动物模型中得到了较好的结果,在人体中仍需研究来进一步证实,而且在不同的个体患者中也表现出不同的应答反应[4-6]。因而,免疫治疗方案只有进行合理的选择,才能使得患者具有最大的获益,而这需要更多的临床证据来证实。
现有的生物标记物并不能很好地解释临床结局的异质性,基因芯片生物信息学技术能够帮助我们更好的了解疾病发生发展的过程,既往研究分析了脓毒症与正常人体之间的基因表达差异性[7-9],然而对于药物干预治疗后引起的变化未得到证实。本研究通过检索基因数据集,获取糖皮质激素用于脓毒症治疗中的基因表达变化,进一步地认识免疫调节相关的网络,从而为免疫治疗在脓毒症患者中的应用提供更多的依据。
从公共数据库(gene expression omnibus,GEO)(https://www.ncbi.nlm.nih.gov/geo/)中通过关键词“sepsis”、“immune”检索并整理出有关脓毒症患者的基因芯片表达数据集:GSE106878,采用Illumina human-6 v2.0 expression beadchip平台,该基因芯片数据集来自外周血标本,包括了24例脓毒症患者接受糖皮质激素治疗前和24例脓毒症患者接受糖皮质激素治疗后的基因表达阵列。
采用R软件及limma、ggplot2、pheatmap软件包对原始数据进行对数标准化处理,按照配对样本来进行表达矩阵的分析比较,再进行差异表达基因的筛选(P<0.05,FC>|logFC均值|+2sd),最终得到糖皮质激素治疗前与治疗后的差异表达基因,取交集后获得上调及下调的差异基因集进行后续的下游分析。
采用R软件及clusterProfiler软件包对脓毒症患者差异表达基因的进行京都基因与基因组百科全书(KEGG,Kyoto Encyclopedia of Genes and Genomes)的通路富集和基因本体论(GO,Gene Ontology)的功能富集。用enrichplot软件包进行基因集富集分析(GSEA,Gene Set Enrichment Analysis)获取基因通路富集信息。
利用基因与蛋白质相互作用检索数据库(STRING-DB,https://string-db.org/)进行基因数据集相关蛋白质的相互作用分析(PPI,protein-proteininteractionnetwork),主要筛选出与免疫、转录、凋亡相关的基因。
经过对糖皮质激素组进行配对数据集的筛选,获得糖皮质激素组的富集分布图,基因存在明显的富集分布(图1),其中上调基因332个,下调基因353个,且上下调基因分布基本均匀(图2)。
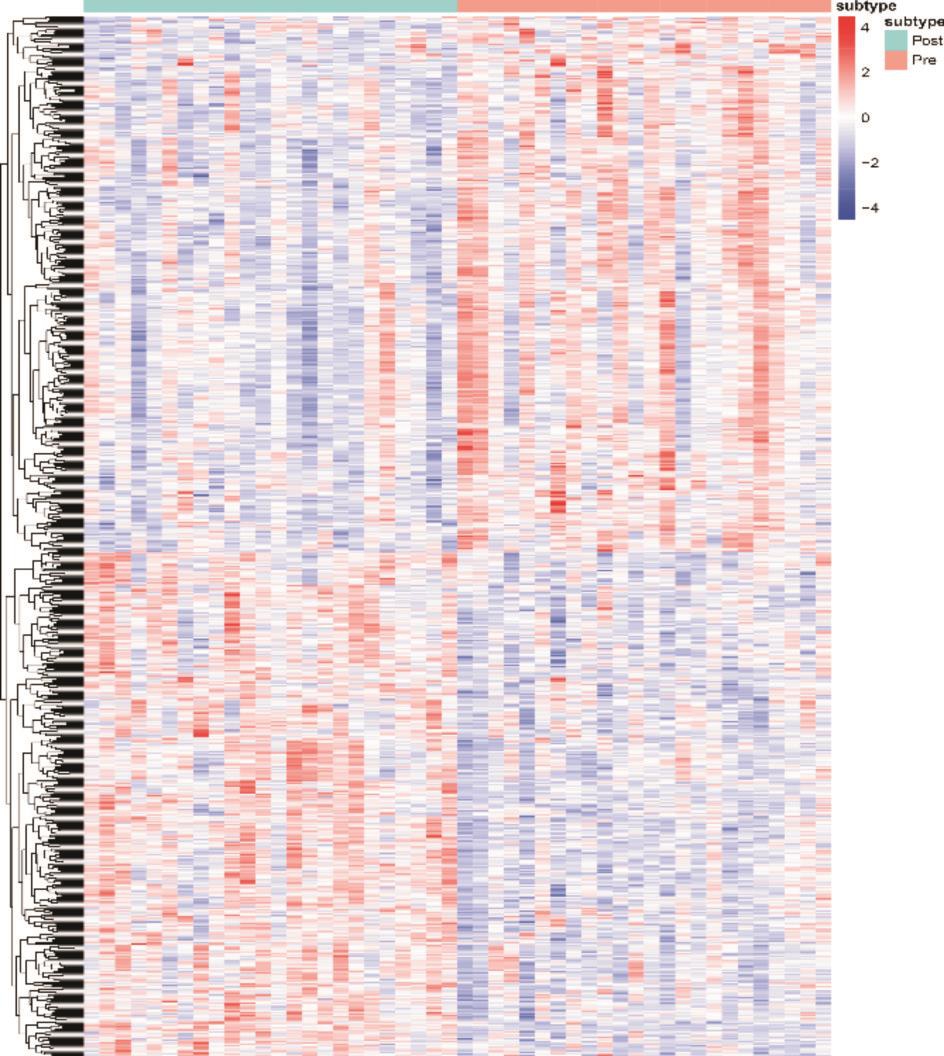
图1 基因富集分布的热图
差异基因进行GO功能富集分析,结果显示,排名前8的生物进程为:①免疫反应中的中性粒细胞激活;②中性粒细胞活化;③中性粒细胞脱颗粒;④中性粒细胞介导的免疫;⑤吞噬作用;⑥白细胞活化的调节;⑦吞噬作用的正向调节;⑧细胞因子产生的正向调控(图3)
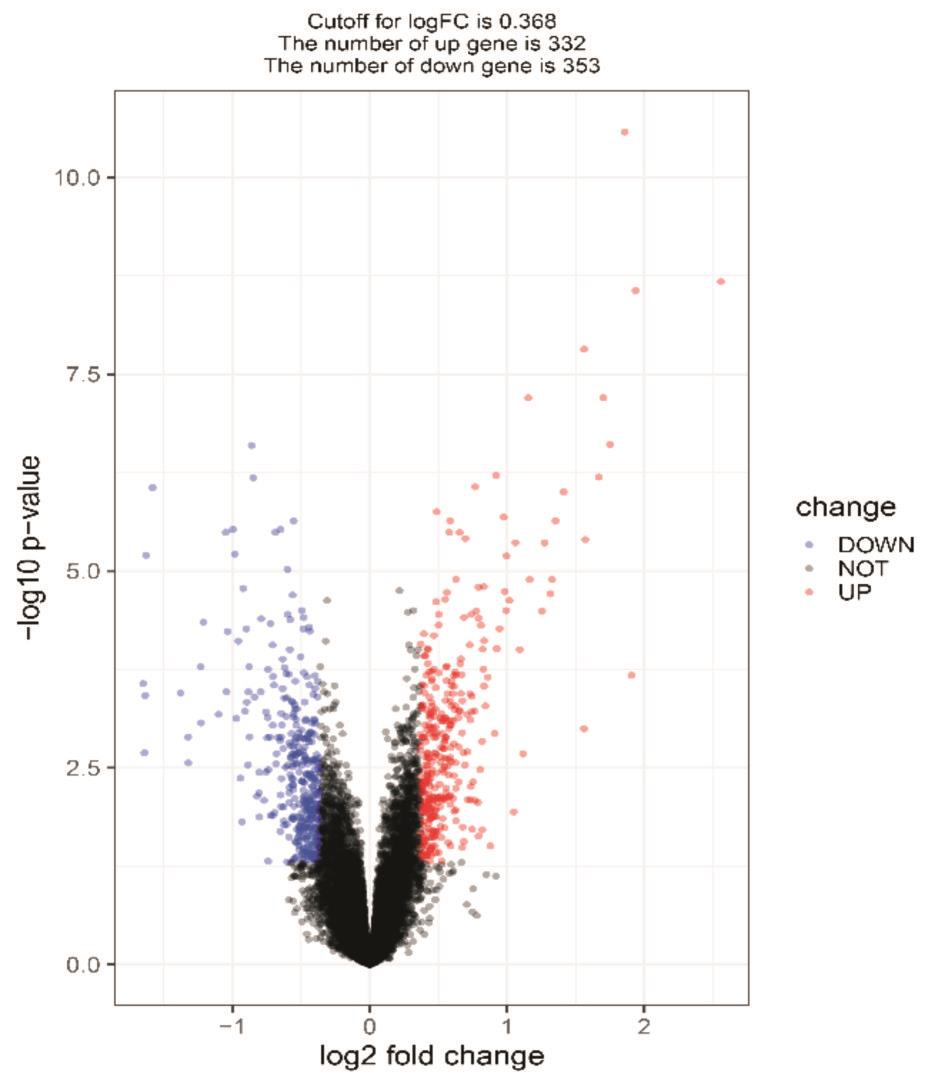
图2 基因富集分布的火山图
差异基因富集的通路主要涉及造血细胞系、吞噬作用、脂代谢等(图4)

图3 GO功能富集
GSEA主要富集的通路有:蛋白质消化与吸
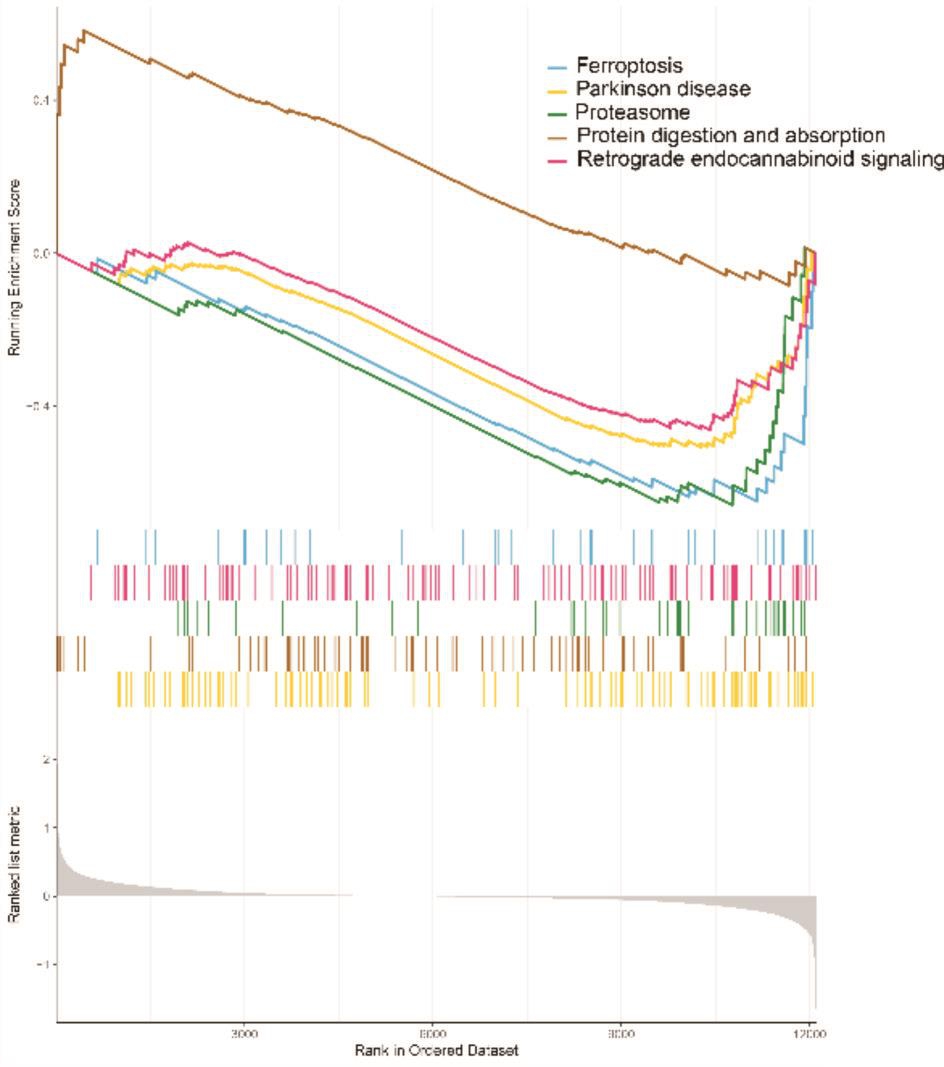
图5 a GSEA富集通路
685个差异基因通过STRING分析后得出免疫应答、粒细胞活化等为主要功能富集(图6a,6b),FPR2、RETN、HMA1、UBB、TFRC、RUMX1等基因编码的蛋白质与多种蛋白存在相互作用关系,在收、药物代谢-细胞色素P450、趋化因子信号通路等(图5a,5b)。网络图中占据了较为核心的位置。
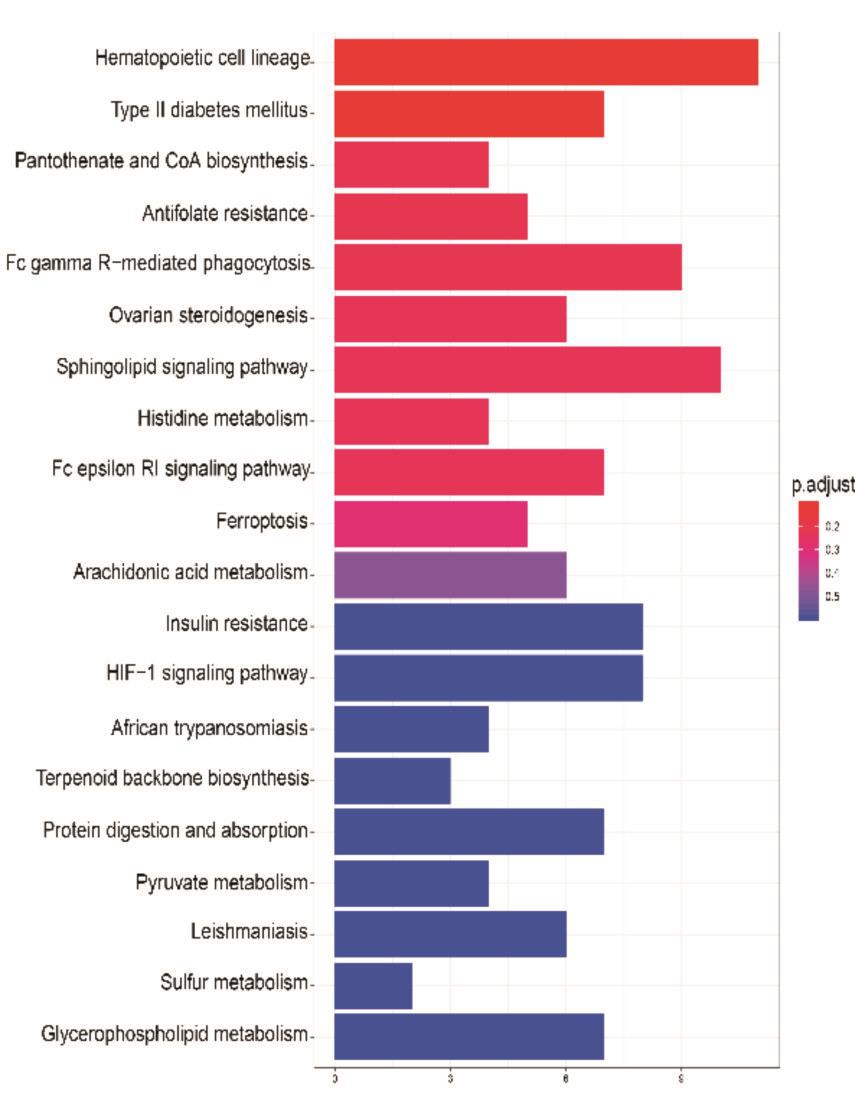
图4 KEGG通路富集
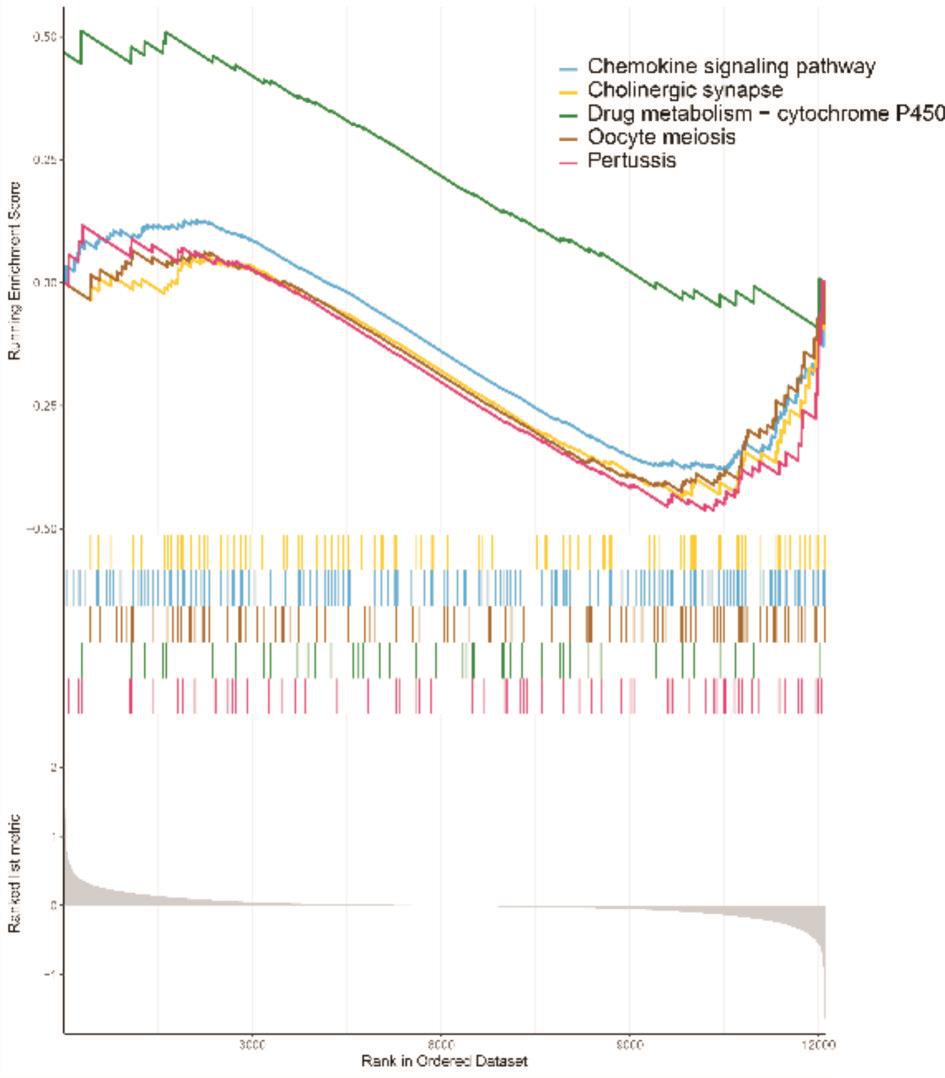
图5 b GSEA富集通路
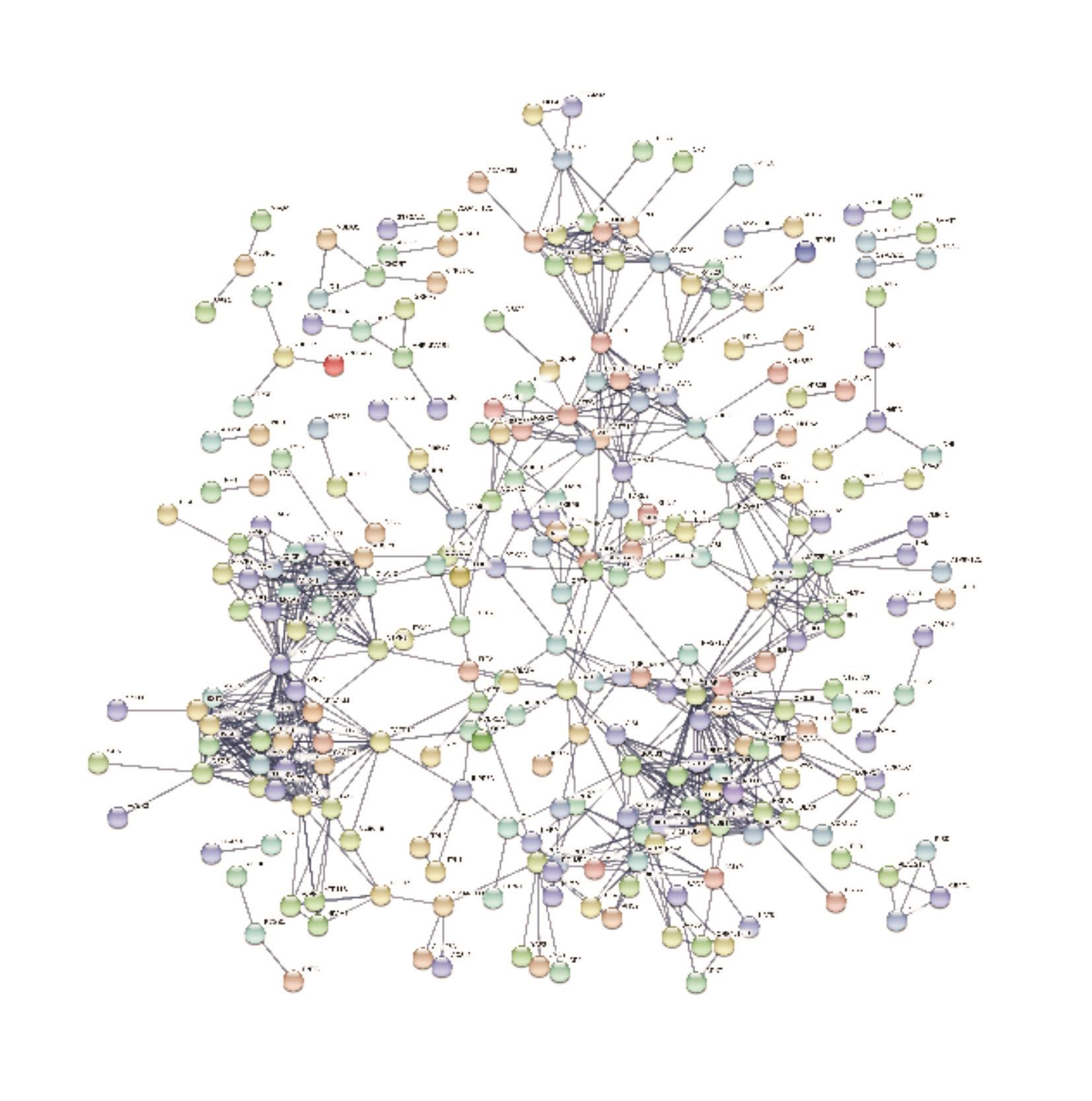
图6 a PPI网络图
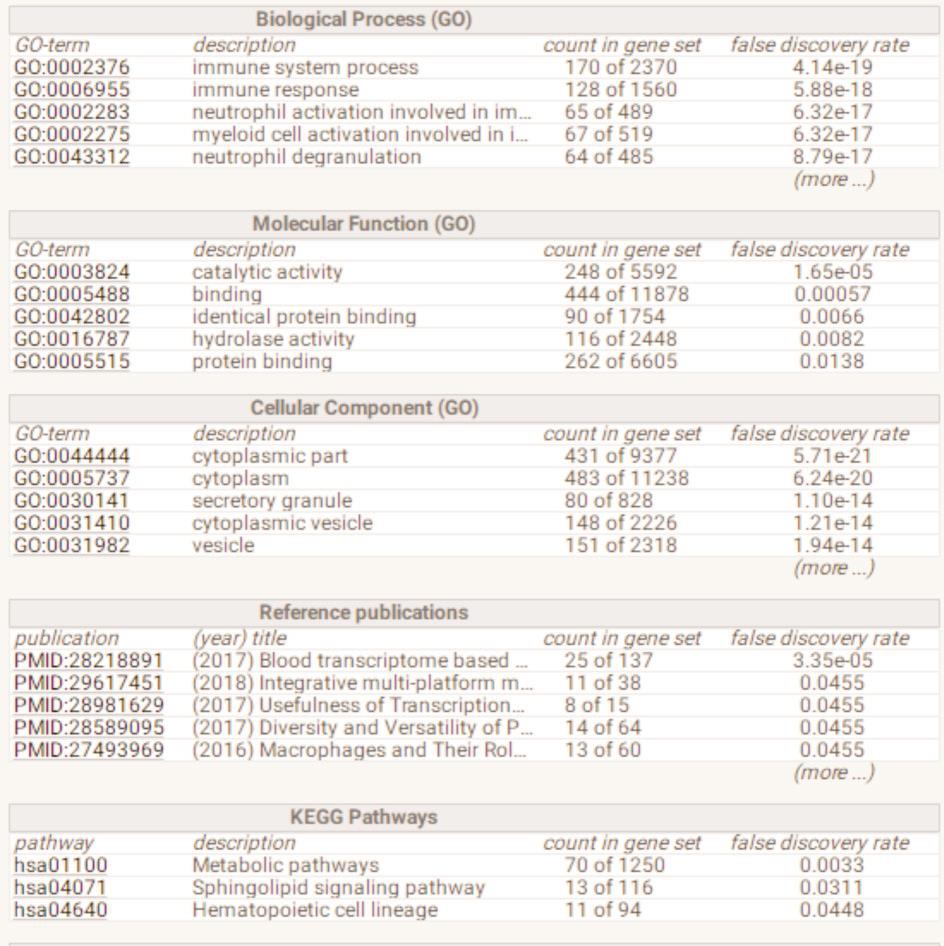
图6 b PPI内关键基因的功能、通路富集
脓毒症是目前临床常见的急危重症疾病,具有复杂的病理生理过程,既往研究表明感染作为初始发病因素,随后激活机体的固有免疫,通过正反馈调节产生级联反应导致了过度炎症反应,后期由于炎症介质的耗竭和免疫细胞的凋亡而进入免疫抑制状态。在炎症反应过度激活与免疫抑制理论的支持下进行了脓毒症的免疫治疗,其中IL-1抗体、IL-6、TNF-α拮抗剂等免疫抑制药物进行早期干预治疗,然而多中心的前瞻性临床试验并未得到理想的结果[10,11]。免疫激活疗法如粒细胞-巨噬细胞集落刺激因子(granulocyte macrophage colony-stimulating factor,GM-CSF)、IFN-γ、IL-7和免疫检查点抑制剂(immune checkpoint inhibitor,ICI),激活患者的免疫潜能有望改善脓毒症患者的临床结局[12-15]。糖皮质激素具有抗炎作用,有双向调节和免疫反应的作用,而糖皮质激素在脓毒症中的应用一直存在争议[16,17]。
目前免疫治疗在脓毒症中的应用多为单药治疗,尚缺乏免疫联合治疗的临床研究,而影响开展免疫联合治疗的原因是多方面的。首先,脓毒症的发生发展的机制仍然不明,治疗后的免疫应答反应也缺乏灵敏度及特异度较高的监测指标,从而致使临床上很难对脓毒症的治疗反应进行有效的监测[18]。其次,目前开展的临床研究得出的研究结论尚无法进行明确的解释,免疫治疗效果在不同患者的结局表现未能得到现有免疫伦理很好的解释。此外,已有的研究表明免疫治疗的联合应用也会带来不良副反应的发生率增加[19],这些原因都给临床上开展脓毒症的免疫联合治疗带来困难。
本研究主要通过分析脓毒症患者经糖皮质激素治疗前后的48例配对样本,进行差异基因的筛选及GO、KEGG、GSEA及PPI功能通路的富集分析,结果主要显示了糖皮质激素参与粒细胞的活化及免疫调节的过程,结合本研究的数据采集于患者接受糖皮质激素治疗后24小时,提示可能参与了脓毒症免疫调节的激活。本研究从基因水平上观察到糖皮质激素在机体免疫调节产生的激活作用,为探索免疫治疗应答的临床生化监测指标提供了可能的思路。本研究由于未能结合转录组、蛋白组、单细胞测序来进一步获取药物在基因表达产生的作用,因而具有一定的局限性。随着生物信息技术的发展,对免疫调节药物作用机理的认识将得到不断加深,为未来脓毒症的免疫联合治疗提供更多的依据。
[1] 中国医师协会急诊医师分会,中国研究型医院学会休克与脓毒症专业委员会[J].中国脓毒症/脓毒性休克急诊治疗指南(2018)[J].中国急救医学,2018,38(9):741-756.
[2] Singer M,Deutschman CS,Seymour CW,et al.The third international consensus definitions for sepsis and septic shock(sepsis-3)[J].JAMA,2016,315(8):801-810.
[3] 赵鹏跃,杜晓辉.脓毒症免疫联合治疗研究进展[J].解放军医学杂志,2019,44(5):434-439.
[4] Sprung CL,Annane D,Keh D,et al.Hydrocortisone therapy for patients with septic shock[J].N Engl J Med,2008,358(2):111-124.
[5] Annane D,Bellissant E,Bollaert PE,et al.Corticosteroids in the treatment of severe sepsis and septic shock in adults:a systematic review[J].JAMA,2009,301(22):2362-2375.
[6] Rice TW,Wheeler AP,Bernard GR,et al.A randomized,double-blind,placebo-controlled trial of TAK-242 for the treatment of severe sepsis[J].Crit Care Med,2010,38(8):1685-1694.
[7] Qin Y,Guo X,Yu Y,et al.Screening key genes and miRNAs in sepsis by RNA-sequencing[J].JChin Med Assoc,2019.doi:10.1097/JCMA.0000000000000209.[Epub ahead of print]
[8] 田阔,王健健,刘佩芳,等.脓毒症患者骨骼肌细胞基因标志物的特征分析[J].中华危重病急救医学,2019,31(3):293-297.
[9] 肖献忠,王慷慨,张华莉,等.基于组学策略的脓毒症研究[J].中国病理生理杂志,2015,(10):1913.
[10]Shakoory B,Carcillo JA,Chatham WW,et al.Interleukin-1 receptor blockade is associated with reduced mortality in sepsis patients with features of macrophage activation syndrome:reanalysis of a prior phase III trial[J].Crit Care Med,2016,44(2):275-281.
[11]Vincent JL,Sun Q,Dubois MJ.Clinical trials of immunomodulatory therapies in severe sepsis and septic shock[J].Clin Infect Dis,2002,34(8):1084-1093.
[12]Meisel C,Schefold JC,Pschowski R,et al.Granulocyte-macrophage colony-stimulating factor to reverse sepsis-associated immunosuppression:a double-blind,randomized,placebo-controlled multicenter trial[J].Am J Respir Crit Care Med,2009,180(7):640-648.
[13]Venet F,Foray AP,Villars-Mechin A,et al.IL-7 restores lymphocyte functions in septic patients[J].J Immunol,2012,189(10):5073-5081.
[14]Watanabe E,Nishida O,Kakihana Y,et al.Pharmacokinetics,pharmacodynamics and safety of nivolumab in patients with sepsis-induced immunosuppression:a multicenter,open-label phase 1/2 study [J]. Shock, 2019.doi: 10.1097/SHK.0000000000001443.[Epub ahead of print]
[15]Hotchkiss R S,Colston E,Yende S,et al.Immune checkpoint inhibition in sepsis:a phase 1b randomized,placebo-controlled,single ascending dose study of antiprogrammed cell death-ligand 1 antibody(BMS-936559)[J].Crit Care Med,2019,47(5):632-642.
[16]Heming N,Sivanandamoorthy S,Meng P,et al.Immune effects of corticosteroids in sepsis[J].Front Immunol,2018,9:1736.
[17]Annane D,Renault A,Brun-Buisson C,et al.Hydrocortisone plus fludrocortisone for adults with septic shock[J].N Engl J Med,2018,378(9):809-818.
[18]Sharma D,Farahbakhsh N,Shastri S,et al.Biomarkers for diagnosis of neonatal sepsis:a literature review[J].JMatern Fetal Neonatal Med,2018,31(12):1646-1659.
[19]Sise ME,Seethapathy H,Reynolds KL.Diagnosis and management of immune checkpoint inhibitor-associated renal toxicity:illustrative case and review[J].Oncologist,2019,24(6):735-742.
Bioinformatics exploration of immunoregulatory mechanism of glucocorticoids in the treatment of sepsis
中图分类号: